Noscendo : Au-delà des panels syndromiques, le « screening total »
Quand votre approche syndromique routinière par PCR n'a rien révélé, que les hémocultures restent négatives et que le traitement empirique anti-infectieux n'améliore pas l'état du patient, que reste-t-il ? Est-ce une impasse ? Noscendo propose un screening plus large, capable de s'affranchir de l'impact des traitements déjà engagés, avec sa solution DISQVERⓇ.
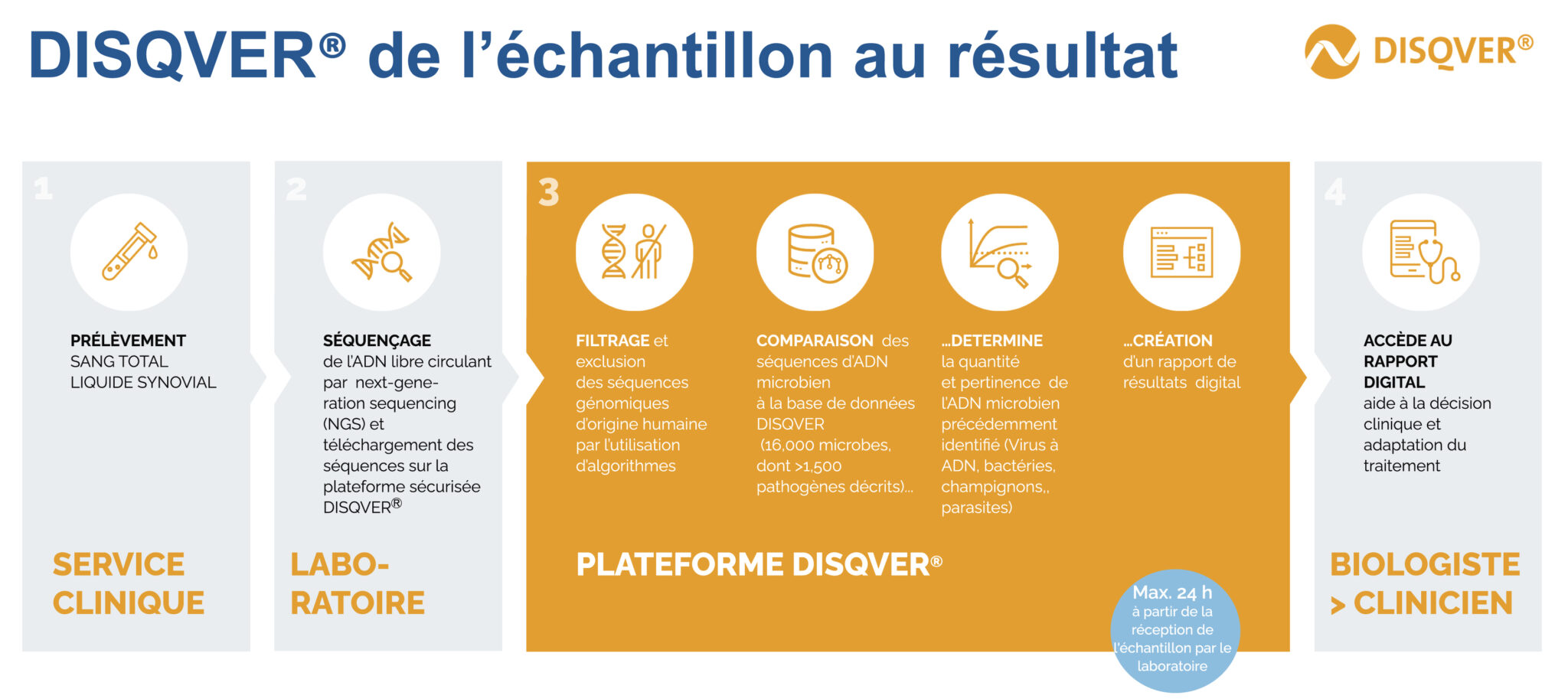
Noscendo - processus disqver3
NoscendoNoscendo est une entreprise d’une cinquantaine de personnes, née en Allemagne en 2017, et construite à partir des travaux des docteurs Silke Grumaz, Philip Stevens et Kai Sohn sur le développement d’une plateforme diagnostique complémentaire pour la détection de microorganismes par séquençage de nouvelle génération (NGS) d’ADN libre circulant à partir d’échantillons de plasma de patients septiques. La solution DISQVER® proposée aujourd’hui est donc née des avancées technologiques récentes : séquençage métagénomique de nouvelle génération et bio-informatique (capacité de calcul et algorithmes améliorés en parallèle d’un travail de fond sur la qualification de la base de données clinique des pathogènes répertoriés).
Diagnostic numérique
 Frédéric Vigier, responsable grands comptes France et Benelux, de Noscendo.DR
Frédéric Vigier, responsable grands comptes France et Benelux, de Noscendo.DR
Ainsi, la solution de Noscendo est une plateforme de diagnostic numérique qui peut identifier 16 000 microorganismes, dont 1 500 pathogènes. Le processus, une fois l’échantillon arrivé au laboratoire, prend 24 heures. « Le résultat généré par comparaison des données brutes à des contrôles de processus et à une population de contrôle non infectée est communiqué au biologiste en nombre de reads normalisés, précise Frédéric Vigier, responsable grands comptes France et Benelux pour la filiale Noscendo en France. Nous sommes les seuls à proposer aujourd’hui une technologie marquée CE-IVD pour la recherche de pathogène sur sang total. »
Si l’utilisation d’ADN libre circulant est aujourd’hui bien développée en oncologie, elle reste encore peu répandue dans le contexte de maladies infectieuses pour la recherche de pathogènes. Ainsi, DISQVER® est une solution bio-informatique permettant d’identifier le ou les pathogènes en cas de coinfection, au niveau de l’espèce, à partir de biopsie liquide (sang, liquide synovial ou lavage bronchoalvéolaire, entre autres). Il s’agit d’une technique agoniste permettant de rechercher aussi bien des bactéries que des champignons, des virus et des parasites.
Pour quelles indications ?

Myriam Livrozet, Market Access Noscendo France / DR
« Aujourd’hui, la métagénomique par séquençage haut débit (mNGS) va au-delà d’une solution de dernier recours, explique Myriam Livrozet, Market Access Noscendo en France. Capable de détecter un large éventail de pathogènes en une seule analyse, sans culture préalable ni influence des traitements en cours, là où les techniques ciblées comme la sérologie ou les tests PCR peuvent être mises en échec. La mNGS représente un atout puissant dans le diagnostic des infections complexes, en cas de pathogènes rares ou émergents, de culture fastidieuse ou non cultivable. Sa rapidité, sa précision et son caractère non invasif en font un complément essentiel aux méthodes traditionnelles, permettant ainsi aux cliniciens d’obtenir un diagnostic exhaustif et fiable pour optimiser la prise en charge des patients. »
Frédéric Vigier ajoute : « Nous sommes aussi pertinents pour des cas où l’approche syndromique n’est pas possible, car il n’y a pas de syndrome évocateur, mais juste une fièvre d’origine inconnue ou une neutropénie fébrile. Nous pouvons alors permettre d’écarter ou non la piste infectieuse et, le cas échéant, orienter vers le bon pathogène. À l’instar de la médecine personnalisée en oncologie, l’identification du pathogène responsable de l’infection permet l’utilisation de l’antimicrobien adapté. »
Des limites ?
« Comme pour n’importe quelle technique diagnostique, il faut en connaitre les limites », souligne Frédéric Vigier. Sa collègue complète : « Notre solution ne donne pas encore d’indication sur l’antibiorésistance, par exemple. Et sur la différenciation entre les espèces colonisatrices ou commensales des espèces à caractère pathogène, nous donnons des indications, mais pour certaines espèces, c’est délicat. Cette indication est construite par des indices de quantification (nombre de reads) – nous avons défini des normes -, mais il incombe au couple clinicien et biologiste de discuter des résultats au vu du contexte clinique. Notre solution bio-informatique gomme cependant beaucoup du bruit de fond grâce à des groupes de contrôle de différents profils déjà implémentés (par exemple, des patients immunodéprimés). »
Données solides, processus encadré
Qu’en est-il de la concurrence ? « Aujourd’hui, beaucoup d’acteurs peuvent faire de la métagénomique. Nos atouts résident dans la solidité et la validité clinique de notre solution, explique Frédéric Vigier. D’une part, notre protocole est rigoureusement encadré – une équipe d’ingénieurs se déplace pour former les équipes afin d’assurer un respect et une maitrise optimale du processus de préparation des échantillons et de génération des données – et, d’autre part, notre base de données est privée et vérifiée, ce qui garantit la fiabilité des résultats. En comparaison, beaucoup d’autres acteurs utilisent des bases cliniques publiques qui ne sont pas toujours à jour et souvent mal documentées, notamment en matière myco- ou parasitologique. Nous avons maintenant des années d’expérience et des milliers d’échantillons dans notre base de données, ce qui nous permet d’affiner encore plus nos procédés et nos méthodes. »
« En France, ajoute Myriam Livrozet, nous sommes heureux d’avoir ainsi été choisis par le département de microbiologie du CHU de Brest, cette année. C’est une vraie marque de confiance et de reconnaissance pour notre solution. »
Des développements en projet
Aujourd’hui présente en Allemagne et en France, Noscendo envisage actuellement une extension au Royaume-Uni.
Pour la France, le cout des actes de métagénomique diagnostique peut être pris en charge dans le cadre du RIHN selon les indications des sociétés savantes.
De son côté, l’entreprise poursuit ses programmes de R&D, en particulier pour explorer la possibilité d’identifier les antibiorésistances, ainsi que ses efforts pour satisfaire à la règlementation CE-IVDR et à l’extension des types d’échantillons validés. « La difficulté réside dans l’obtention de suffisamment d’échantillons pour valider les études donnant accès à l’IVDR, précise Myriam Livrozet. Le passage à l’IVDR est un défi financier en raison du nombre d’études à mener pour soumettre le dossier. »
L’entreprise reste également attentive aux nouveautés techniques en matière de séquençage et aux nouveaux équipements mis sur le marché.
Quelle place dans le parcours clinicobiologique ?
Dernier recours ou accès direct ? Dans certains cas, serait-il plus judicieux de passer directement par la métagénomique ? « C’est un choix médicoéconomique et clinicobiologique qui, pour l’instant, n’est pas encore évalué, la solution n’étant pas assez démocratisée pour mener ce genre d’étude », détaille Myriam Livrozet. Cependant, on note que certaines spécialités montrent un intérêt grandissant pour cette approche. Ainsi, la société internationale des maladies infectieuses cardiovasculaires (ISCVID) a intégré le recours à la mNGS dans ses critères de Duke 2023, pour le diagnostic des endocardites infectieuses.
 Se connecter
Se connecter