La virulence du staphylocoque doré ne tient qu'à un nucléotide
Dirigée par une équipe du CNRS des chercheurs ont mis en évidence une nouvelle enzyme (méthyltransférase RlmQ) dont l'inactivation engendre des modifications sur l'ARN ribosomal, sur la traduction et sur la virulence de Staphylococcus aureus.
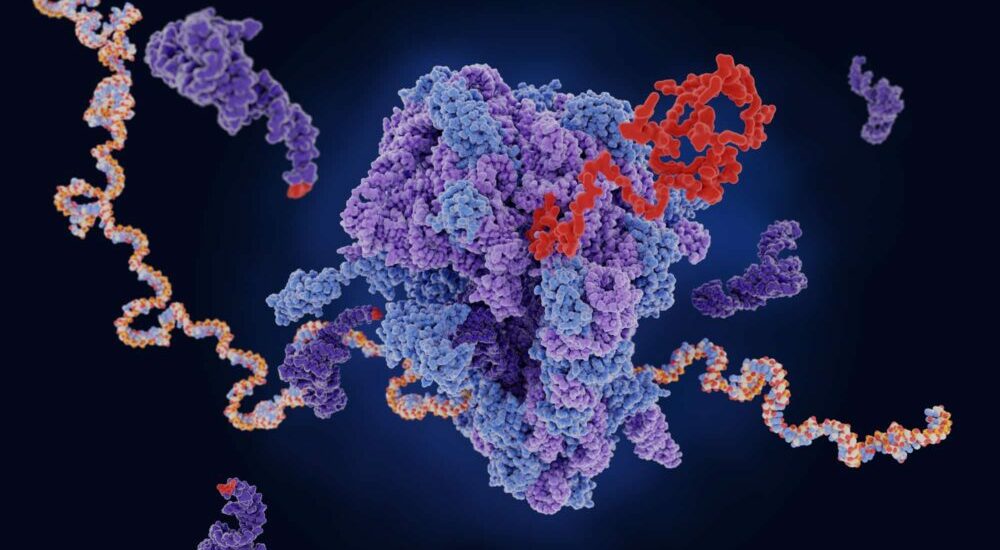
Publiés dans la revue RNA, les résultats de recherche dirigée par l’équipe CNRS du département « Architecture et réactivité de l’ARS » de l’Université de Strasbourg, dévoilent une nouvelle enzyme – la méthyltransférase RlmQ – impliquée dans des modifications sur l’ARN ribosomal (ARNr) des bactéries Gram positif – comme Staphylococcus aureus. Cette enzyme agit lors de la biogenèse du ribosome en modifiant spécifiquement un seul nucléotide de l’ARNr en lui ajoutant un groupement méthyl.
La composition de l’ARNr au coeur de la traduction
On sait aujourd’hui que des modifications de l’ARNr peuvent entraîner des changements importants dans la vitesse et la précision de la traduction des ARN messagers (ARNm) en protéines. Cependant les mécanismes sous-jacent et l’impact des modifications restent aujourd’hui souvent insaisissables et imprévisibles. Pour tenter de percer certains de ces mécanismes des études sont en cours, notamment en comparant des modifications de l’ARNr dans des groupes de bactéries éloignées sur le plan taxonomique. Cette comparaison permet de cerner plus facilement le rôle général ou spécifique de certaines modifications
Gram positif Vs. gram négatif
Dans l’étude publié dans la revue RNA, les chercheurs ont ainsi étudié en comparaison une bactérie Gram positif (Staphylococcus aureus) et une bactérie Gram négatif (E.coli). Ils ont ainsi identifié une nouvelle méthyltransférase, RlmQ, chez Staphylococcus aureus, responsable de la modification spécifique m7G2601 chez les bactéries Gram positives, qui n’est pas modifiée chez Escherichia coli (G2574). Par ailleurs, ils ont aussi identifiés l’absence de méthylation sur C1989 (qui est l’équivalent à C1962 chez E. coli et qui, elle, est méthylée en position 5 par la méthyltransférase RlmI, un paralogue de RlmQ, spécifique des Gram négatifs).
Petites modifications, grands impacts
Les chercheurs ont remarqué que les deux modifications (S. aureus m7G2601et E. coli m5G2574) sont positionnées dans le corridor du ribosome que parcourent les ARN de transfert (ARNt). Une place stratégique donc. Ils supposent alors que ces modifications ont une fonction commune potentielle dans la traduction. En inactivant RlmQ chez S. aureus ils observent la perte de la méthylation au niveau de G2601 avec un impact significatif sur la croissance, la cytotoxicité et la formation de biofilms. Ces résultats dévoilent les liens complexes entre les modifications de l’ARNr, la traduction et la virulence chez les bactéries pathogènes à Gram positif.
Une meilleure compréhension de ces mécanismes pourrait ouvrir de nouvelles voies pour le développement de thérapies ciblées contre les infections bactériennes, offrant ainsi des perspectives passionnantes pour la recherche future dans le domaine de la microbiologie.
En savoir plus :
Bahena-Ceron R, Teixeira C, Ponce JRJ, Wolff P, Couzon F, François P, Klaholz BP, Vandenesch F, Romby P, Moreau K, Marzi S. RlmQ: a newly discovered rRNA modification enzyme bridging RNA modification and virulence traits in Staphylococcus aureus. RNA. 2024 Feb 16;30(3):200-212. doi: 10.1261/rna.079850.123.


